Семинары 2020/2021
08.12.2021 [online] Метод доменной адаптации для получения аннотации G-квадруплексами генома мыши
Докладчик: Латышев Павел, стажер-исследователь
Аннотация генома устойчивыми G-квадруплексами, которые располагаются на хроматине клетки, полученная в результате метода G4-ChIP-Seq есть только для человеческого генома. В данном докладе будет представлен метод аннотации генома мыши G-квадруплексами с использованием метода DANN (Domain-Adversarial Training of Neural Networks) из класса unsupervised domain adaptation. Было проведено сравнение наивного применения модели, обученной на данных человека, на геноме мыши и модели, полученной с использованием DANN, в результате число ложноположительных срабатываний снизилось с 61% до 49%.
24.11.2021 [online] Методы интерпретации при помощи omics-данных в задачах предсказания Z-ДНК
Докладчик: Бекназаров Назар, младший научный сотрудник
Проблема предсказания Z-DNA была неразрешенной долгое время. Было понимание что использование только последовательности ДНК не достаточно чтобы решить эту проблему. DeepZ решает проблему предсказания Z-DNA используя методы машинного обучения и дополнительные омиксные данные. Данный подход не только решил задачу предсказания Z-DNA, но и открывает широкие перспективы для дальнейшего применения ранее не решенных задач
17.11.2021 [online] Методы глубинного обучения для предсказания G-квадруплексов с использованием омиксных данных
Докладчик: Латышев Павел, стажер-исследователь
В настоящее время не существует вычислительных методов, способных предсказывать с высоким качеством образование устойчивых G-квадруплексов на хроматине. В данном докладе предлагается метод предсказания формирования G-квадруплексов с использованием нейронной LSTM-сети. Также будет рассмотрено использование омиксных данных из ChIP-Atlas для предсказания данных структур, что дало увеличение качества распознавания. Кроме того, будет предложен метод поиска значимых эпигенетических признаков, связанных с образованием G-квадруплексов.
03.11.2021 [online] Поиск консервативных вторичных структур в вирусах семейства Coronaviridae
Докладчик: Шеин Александр, младший научный сотрудник
Вирусы семейства Coronaviridae являются обширным семейством РНК-содержащих вирусов, инфицирующих значительное число видов позвоночных, включая птиц, рептилий и млекопитающих. В свете событий последних двух лет, исследование данного семейства вирусов, и, SARS COV2 в частности, является очень актуальной задачей. Одним из перспективных направлений исследований является поиск консервативных вторичных структур в геномных последовательностях вирусов, предположительно влияющих на такие процессы, как репликация и заражение клеток хозяина. В данной работе представлен анализ вторичных структур РНК у 39 различных видов коронавирусов. В ходе работы были обнаружены консервативные структуры стебель-петля нескольких геномных локациях: в кодирующих областях ORF1a и b, Surface Glycoprotein и области ORF7. Дальнейший анализ найденных структур, в частности, в белке S может позволить лучше понять жизненный цикл вируса и механизмы его взаимодействия с клеткой-хозяином.
27.10.2021 [online] Кластеризация и описание IFNb-регулируемых генов
Докладчик: Федоров Александр, младший научный сотрудник
Интерфероны (IFN) - это ряд сигнальных белков со схожими свойствами, синтезируемых клеткой при распознавании Damage Associated Molecular Patterns (DAMPs) и/или Pathogen Associated Molecular Patterns (PAMPs). Согласно классическому представлению, данные белки необходимы для стимуляции клеточного иммунитета, например, экспрессии белков, препятствующих репликации вирусов. Данный взгляд, однако, не совсем корректен для IFN бета (IFNb), что мы смогли показать используя публичные Total RNA-seq эксперименты для IFNb-стимулированных мышиных фибробластов. Конкретно, после 48 часов стимуляции почти половина всей мРНК в клетке не относится к известным генам иммунной системы, а принадлежит к конкретным факторам трансляции или рибосомальным белкам. Только часть из данных факторов была ранее ассоциирована со стрессовым состоянием клетки или иммунным ответом.
13.10.2021 [online] Образование Z-РНК в ответ на вирусную инфекцию
Докладчик: Коновалов Дмитрий, стажер-исследователь
Каждый человек на протяжении своей жизни сталкивается с вирусами гриппа и герпеса. Было показано, что реплицирующийся вирус генерирует Z-РНК, которые активируют ZBP1 в ядрах инфицированных клеток, что в конечном итоге вызывает клеточную смерть. Исследование механизмов ответа на инфекцию может помочь в борьбе с этими заболеваниями. В данной работе представлен анализ экспрессии генов, образующих Z-РНК через 8 и 12 часов после инфекции эмбриональных фибробластов мыши вирусом гриппа типа А и вирусом герпеса первого типа.
22.09.2021 [online] Анализ редактирования РНК: краткий обзор
Докладчик: Федоров Александр, младший научный сотрудник
Редактирование РНК - это один из способов посттранскрипционной регуляции экспрессии генов, важный механизм клеточного иммунитета в эукариотических клетках. В докладе будут описаны основные “редакторы” РНК в клетках мыши и человека, белки семейств ADAR и APOBEC, текущее представление о роли A-I редактирования в регуляции иммунного ответа. Дано подробное описание существующих вычислительных подходов к анализу редактирования на уровне отдельных локусов, регионов, всего генома. Описаны их основные преимущества, ограничения, приведены типичные варианты применения.
28.05.21 [online] Математическое моделирование этнических генотипов
Докладчик: Александра Глотова, студентка 4 курса бакалавриата МИЭМ
Этническая принадлежность играет важную роль в предрасположенности члена группы к развитию определенного заболевания и реакции на определен ную дозу лекарства. В данной работе представлен анализ фармакогенетиче ского тестирования этнических групп Российской Федерации с использованием различных методов машинного обучения. Также в ходе работы была модифици рована и предсказана допустимая доза варфарина для рассматриваемых групп, определена вероятность повторного инсульта у пациентов, принимающих кло пидогрел. Данное исследование может помочь расширить знания о представите лях этнических групп России, уменьшить риск побочных реакций на различные препараты.
21.05.21 [online] Применение генеративно-состязательных сетей к задачам распознавания вторичных структур ДНК
Докладчик: Никита Казаков, студент 4 курса бакалавриата МИЭМ
ДНК – одна из важнейших макромолекул любого организма. Она состоит из последовательностей нуклеотидов и отвечает за хранение информации, передачи ее из поколения в поколение, а также за многие другие функции в клетках живых организмов. Активное изучение ДНК началось еще в конце 19 столетия [1] и по настоящее время проблема не теряет своей актуальности. Особый интерес для исследователей имеют вторичные структуры ДНК, которые представляют собой расположение молекулы ДНК в пространстве, определяющее и саму структуру ДНК и её функции. У структуры Z-ДНК, в отличие от самых распространённых структур А и В, спираль закручена влево. Точные закономерности построения данной структуры в настоящий момент неизвестны, а количество экспериментально полученных данных мало, что мешает корректно обучать некоторые модели машинного обучения. Именно поэтому распознавание и генерация Z-ДНК при помощи нейронных сетей является важной задачей в исследовании данной структуры. Работа посвящена применению генеративно-состязательных нейронных сетей к задачам распознавания вторичных структур ДНК, в частности Z-ДНК. Задача распознавания Z-ДНК это одна из важнейших задач, поскольку ее решение позволяет определять является ли данная последовательность нуклеотидов закрученной влево спиралью ДНК или нет. Задача генерации – создать последовательности нуклеотидов, при которых ДНК имело бы структуру Z-ДНК. Созданные последовательности можно использовать с целью улучшения качества других моделей, которые, например, требуют большой массив данных. В результате данной работы были протестированы и улучшены некоторые структуры нейронных сетей, которые в дальнейшем распознают Z-ДНК с точностью до 1.0, и генерируют последовательности, которые распознаются CNN с точностью до 1. Дополнительно, перед распознаванием и генерацией Z-ДНК разработанные нейронные сети были протестированы на промоторных областях ДНК человека. Данная работа является одной из первых работ по использованию нейронных сетей в задачах генерации и распознавания Z-ДНК.
16.04.21 [online]
Докладчик: Иван Антонов, н.с. лаборатории биоинформатики
По материалам публикации The transcriptional regulator Aire binds to and activates super-enhancers (Bansla et al. Nat Immunol
. 2017 Mar;18(3):263-273. doi: 10.1038/ni.3675.)
Aire is a transcription factor that controls T cell tolerance by inducing the expression of a large repertoire of genes specifically in thymic stromal cells. It interacts with scores of protein partners of diverse functional classes. We found that Aire and some of its partners, notably those implicated in the DNA-damage response, preferentially localized to and activated long chromatin stretches that were overloaded with transcriptional regulators, known as super-enhancers. We also identified topoisomerase 1 as a cardinal Aire partner that colocalized on super-enhancers and was required for the interaction of Aire with all of its other associates. We propose a model that entails looping of super-enhancers to efficiently deliver Aire-containing complexes to local and distal transcriptional start sites.
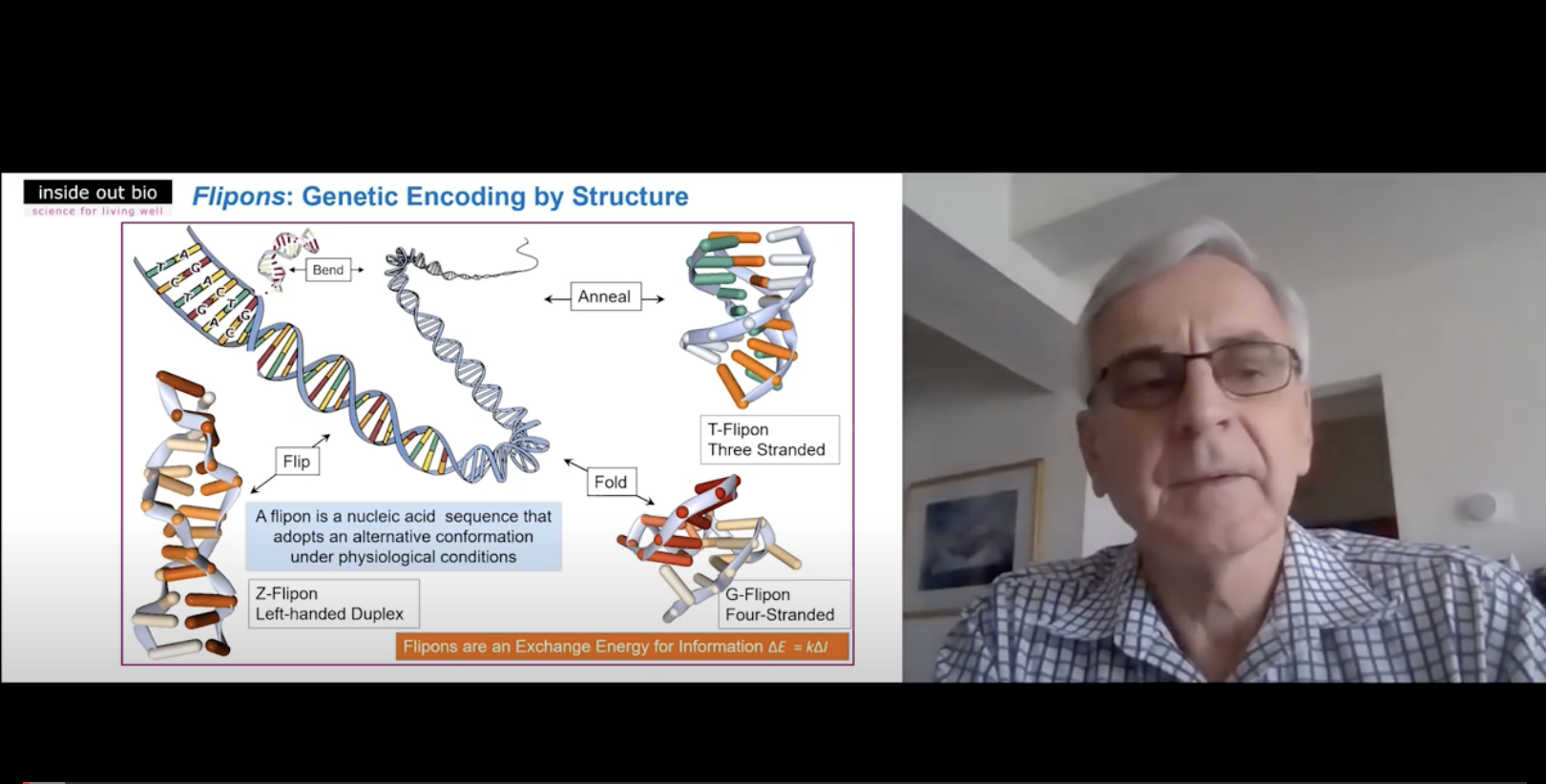
13.04.21 Genetic Computers
Faculty of Computer Science Colloquium "Genetic Computers "
Speaker: Alan Herbert,
InsideOutBio; HSE University.
Abstract:
• Biologists and Engineers view Programming Problems very Differently
• Encoding of Genetic Information is by both Nucleic Acid Structure (flipons) and Sequence (codons)
• Encoding Genetic Programs by Structure is More Adaptive than by Sequence
• I will use the Biology of Left-handed Z-DNA and Z-RNA to Exemplify these Principles
• I will also Describe the Potential Use of Other Flipon Classes in Genetic Programming
09.04.21 Fully bioresorbable, leadless, battery-free cardiac pacemake
Zoom
Presenter: Igor Efimov, PhD, FNAI, FAIMBE, FAHA, FHRS Alisann and Terry Collins Professor, Department of Biomedical Engineering The George Washington University, Washington DC Editor-in-Chief, Cardiovascular Engineering & Technology
Temporary cardiac pacemakers provide critical functions in pacing through periods of need during post-surgical recovery. The percutaneous leads and externalized hardware associated with these systems present, however, risks of infection and constraints on patient mobility. Furthermore, the pacing leads can become enveloped in fibrotic tissue at the electrode-myocardium interface, which thereby increases the potential for myocardial damage and perforation during lead removal. Here, we report a bioresorbable, leadless, and fully implantable cardiac pacemaker for post-operative control of cardiac rate and rhythm during a stable operating timeframe that subsequently undergoes complete dissolution and clearance via natural biological processes. A combined set of in vitro, ex vivo, and in vivo studies across mouse, rat, rabbit, canine, and human cardiac models demonstrates that these devices provide an effective, battery-free means for pacing hearts of various sizes with tailored geometries and timescales for operation and bioresorption. These features enable programmable cardiac pacing in a manner that overcomes all of the key disadvantages of traditional temporary pacing devices. As such, this novel cardiac pacemaker may serve as the basis for the next generation of post-operative temporary pacing technology.
Professor Efimov is the Director of Cardiovascular Engineering Laboratory, an NIH-funded cardiovascular research and engineering laboratory, which focusses on the physiological mechanisms of cardiovascular disease and on development of novel therapies for heart diseases with emphasis on heart rhythm disorders. Professor Efimov served as an inaugural chairman of Biomedical Engineering Department at the George Washington University from January 2015 to December 2019. In 2008, Dr. Efimov founded Cardialen to develop low energy implantable electrotherapy for cardiac arrhythmias. Cardialen has raised over $30M in venture and federal funding and is conducting clinical trials worldwide. In 2020, Dr. Efimov co-founded NuSera Biosystems to develop new catheter technology for mapping and ablation of arrhythmias.
Dr. Efimov earned his M.Sc. and PhD from Moscow Institute of Physics and Technology in 1986 and 1992, respectively, and completed his postdoctoral training at the University of Pittsburgh (1992-1994). He served on the faculty of the Cleveland Clinic Foundation (1994-2000) and Case Western Reserve University (2000-2004) in Cleveland, OH, and Washington University in St. Louis, MO (2004-2015), prior to joining the George Washington University in 2015 as the founding chairman of its new BME Department.
Dr. Efimov is Fellow of National Academy of Inventors, the American Institute for Medical & Biological Engineering, the Heart Rhythm Society, the American Heart Association, etc. He has received numerous awards including: 2021 HRS Distinguished Scientist Award, RASA George Gamow Prize, Astor Visiting Fellowship at University of Oxford, Washington University Chancellor’s Hartwell Prize for Innovative Research, The Doris J.W. Escher Lectureship at Montefiore Medical Center of the Albert Einstein College of Medicine, Walter Lillehei Lectureship at University of Minnesota, Richard B. and Lynne V. Cheney Lectureship at the George Washington University, etc. Dr. Efimov held visiting professorships at the University of Bordeaux, France; University of Nizhny Novgorod and Moscow Institute of Physics and Technology, Russia.
02.04.21 [online] Молекулярные механизмы онкогенеза через призму нуклеосом и гистонов
Докладчики: Анна Грибкова, Гупта Шабханги, Алексей Шайтан, к.ф.-м.н., научный сотрудник международной лаборатории биоинформатики факультета компьютерных наук, ведущий научный сотрудник, кафедра биоинженерии, биологический факультет, МГУ им. М.В. Ломоносова
Доклад будет посвящен механизмам развития и прогрессирования онкологических заболеваний, опосредованных гистонами и нуклеосомой. Будет рассмотрено влияние гистоновых мутаций и изменений экспрессии гистонов на эпигенетические процессы канцерогенеза. Особое внимание будет уделено гистоновым вариантам и изоформам (как белковым сплайс-изоформам, так и каноническим). Также будут рассмотрены факторы, влияющие на скорость мутаций: геометрические и стерифеские эффекты компактизации ДНК в нуклеосомах и взаимодействие с транскрипционными факторами, включая пионерные.
26.03.21 [online] ZBP1: врожденный сенсор, регулирующий гибель и воспаление клеток
Докладчик: Мария Попцова, заведующая международной лабораторией биоинформатики
По материалам публикации Kuriakose and Kanneganti, Trends Immunol. 2018 February ; 39(2): 123–134
Z-ДНК-связывающий белок 1 (ZBP1), первоначально описанный как IFN-индуцируемый, ассоциированный с опухолью белок, содержит домены связывания нуклеиновых кислот с левозакрученной формой спирали (Z-формы) и домены RHIM для гомотипических взаимодействий белков. Недавние исследования определили ZBP1 как врожденный сенсор вирусных инфекций и мишень для стратегии уклонения от вирусов, регулирующих гибель клеток, активацию инфламмасом и провоспалительные реакции. ZBP1 также активен во время развития организма и может вызывать перинатальную летальность, если его RHIM-зависимые взаимодействия не ограничены. В обзоре обсуждается роль ZBP1 в качестве сенсора патогена и центрального регулятора гибели клеток и воспалительных реакций. Также обсуждаются открытые вопросы регуляторных функций ZBP1 и намечаются потенциальные направления для будущих исследований.
19.03.21 [online] Интегративное моделирование структуры и динамики биомакромолекулярных комплексов
Докладчик: Алексей Константинович Шайтан, к.ф.-м.н., научный сотрудник международной лаборатории биоинформатики факультета компьютерных наук, ведущий научный сотрудник, кафедра биоинженерии, биологический факультет, МГУ им. М.В. Ломоносова
Доклад будет сделан по материалам представленной к защите диссертации на соискание степени доктора физико-математических наук по специальности 03.01.09 Математическая биология, биоинформатика.
В докладе будет рассказано о применении методов компьютерного моделирования для установления структуры и изучения больших комплексов биомолекул. В частности будет рассказано об использовании суперкомпьютерных расчетов методом молекулярной динамики для изучения нуклеосом - элементарных единиц упаковки ДНК в геномах эукариот. Будет рассказано о том, каким образом, комбинируя методы молекулярного моделирования с различными косвенными экспериментальными данными, можно изучать упаковку ДНК в ядре клетки и структуру амилоидных фибрилл.
12.02.21 [online] Genome-wide analysis of DNA G-quadruplex motifs across 37 species provides insights into G4 evolution
Докладчик: Юлия Терентьева (стажер-исследователь МЛ биоинформатики).
Доклад по материалам публикации Wu, F., Niu, K., Cui, Y. et al. Commun Biol 4, 98 (2021).
Genome-wide analysis of DNA G-quadruplex motifs across 37 species provides insights into G4 evolution G-quadruplex (G4) structures have been predicted in the genomes of many organisms and proven to play regulatory roles in diverse cellular activities. However, there is little information on the evolutionary history and distribution characteristics of G4s. Here, whole-genome characteristics of potential G4s were studied in 37 evolutionarily representative species. During evolution, the number, length, and density of G4s generally increased. Immunofluorescence in seven species confirmed G4s’ presence and evolutionary pattern. G4s tended to cluster in chromosomes and were enriched in genetic regions. Short-loop G4s were conserved in most species, while loop-length diversity also existed, especially in mammals. The proportion of G4-bearing genes and orthologue genes, which appeared to be increasingly enriched in transcription factors, gradually increased. The antagonistic relationship between G4s and DNA methylation sites was detected. These findings imply that organisms may have evolutionarily developed G4 into a novel reversible and elaborate transcriptional regulatory mechanism benefiting multiple physiological activities of higher organisms.
05.02.21 [online] Разнообразие биологии РНК: от запрограммированных сдвигов рамки считывания до длинных некодирующих РНК
Антонов Иван Валентинович, PhD, научный сотрудник Международной лаборатории биоинформатики, НИУ ВШЭ, доцент Факультета компьютерных наук, НИУ ВШЭ, научный сотрудник Института Биоинженерии, ФИЦ ФОБ РАН
В своем докладе Иван Антонов представит краткий обзор проектов по различным проектам, направленным на изучение роли РНК в молекулярной биологии. Он отразил основные результаты, полученные им в за более чем 10 летний стаж работы в этой области. В первой части доклада рассказано о новом алгоритме предсказания межмолекулярных РНК-РНК взаимодействий ASSA, а также о применении этого алгоритма к данным проекта FANTOM6. В результате этой работы было найдено более 10 длинных некодирующих РНК (нкРНК), которые могут регулировать экспрессию других генов в ядре клеток человека, связываясь с ними ко-транскрипционно. Также рассказано о крупномасштабном анализе предсказанных РНК-ДНК триплексов, результатом которого стало выявление так называемых "универсальных сайтов связывания через триплексы". Наконец, будут представлены предварительные результаты по изучению взаимодействия между туберкулезной бактерией и макрофагом, которого она заражает. Вторая часть доклада будет посвящена открытию новых случаев запрограммированного сдвига рамки считывания (СРС) в генах прокариот. Для выявления этих случаев была разработана программа GeneTack для ab initio предсказания генов с СРС. С помощью GeneTack были предсказаны все гены с потенциальными СРС в более чем 1000 прокариотических геномах и с помощью методов сравнительной геномики были выделены наиболее перспективные кандидаты для последующей экспериментальной проверки. Наиболее интересные экспериментальные результаты были получены для гена магний/кобальт хелатазы chlD, который был детально изучен в отдельном анализе.
29.01.21 [online] Extensive editing of cellular and viral double- stranded RNA structures accounts for innate immunity suppression and the proviral activity of ADAR1
Докладчик: Назар Бекназаров (стажер-исследователь МЛ биоинформатики).
Доклад по материалам публикации
Citation: Pfaller CK, Donohue RC, Nersisyan S, Brodsky L, Cattaneo R (2018) Extensive editing of cellular and viral double-stranded RNA structures accounts for innate immunity suppression and the proviral activity of ADAR1p150. PLoS Biol 16(11): e2006577
The interferon (IFN)-mediated innate immune response is the first line of defense against viruses. However, an IFN-stimulated gene, the adenosine deaminase acting on RNA 1 (ADAR1), favors the replication of several viruses. ADAR1 binds double-stranded RNA and converts adenosine to inosine by deamination. This form of editing makes duplex RNA unstable, thereby preventing IFN induction. To better understand how ADAR1 works at the cellular level, we generated cell lines that express exclusively either the IFN-inducible, cytoplasmic isoform ADAR1p150, the constitutively expressed nuclear isoform ADAR1p110, or no isoform. By comparing the transcriptome of these cell lines, we identified more than 150 polymerase II transcripts that are extensively edited, and we attributed most editing events to ADAR1p150. Editing is focused on inverted transposable elements, located mainly within introns and untranslated regions, and predicted to form duplex RNA structures. Editing of these elements occurs also in primary human samples, and there is evidence for cross-species evolutionary conservation of editing patterns in primates and, to a lesser extent, in rodents. Whereas ADAR1p150 rarely edits tightly encapsidated standard measles virus (MeV) genomes, it efficiently edits genomes with inverted repeats accidentally generated by a mutant MeV. We also show that immune activation occurs in fully ADAR1-deficient (ADAR1KO) cells, restricting virus growth, and that complementation of these cells with ADAR1p150 rescues virus growth and suppresses innate immunity activation. Finally, by knocking out either protein kinase R (PKR) or mitochondrial antiviral signaling protein (MAVS)—another protein controlling the response to duplex RNA—in ADAR1KO cells, we show that PKR activation elicits a stronger antiviral response. Thus, ADAR1 prevents innate immunity activation by cellular transcripts that include extensive duplex RNA structures. The trade-off is that viruses take advantage of ADAR1 to elude innate immunity control.
Время проведения: 18:00-19:30.
20.11.20 [online] Поиск пиков в ChIP-seq данных: обзор современных методов и подходов
Докладчик: Александр Федоров (стажер-исследователь МЛ биоинформатики)
Ключевой стадией обработки ChIP-seq экспериментов является поиск областей, обогащенных прочтениями относительно контроля. Простота данной задачи обманчива, она не имеет формального определения, а общепринятого подхода к ее решению не существует. Мы рассмотрим основные проблемы обработки СhIP-seq данных, идеи, лежащие в основе популярных алгоритмов, а также затронем проблему выбора метрик оценки качества.
Время проведения: 18:00-19:30.
06.11.20 [online] The role of G-quadruplexes in differential methylation
Докладчик: Дмитрий Коновалов (преподаватель ФКН, аспирант физического факультета МГУ им Ломоносова)
Currently it is not fully understood how methylation level affects stability and dynamic of quadruplexes (G4s). Quadruplexes were found both in hypo- and hypermethylated regions. It was shown that G4s are associated with hypomethylation in the human genome, and it is thought that G4 locally regulates methylation level by binding and inhibiting DNMT1. On the other hand methylation induces formation of quadruplexes with increased stability that can arrest DNA polymerase. Here we explored distribution of quadruplexes with different types of differentially methylated positions (DMPs or DMCs) including associated wth sex, age, tissue type and cell differentiation. We found statistically significant depletion of quadruplexes in hypermethylated DMCs in all types of DMCs. The 28-fold enrichment of quadruplexes in hypomethylated DMC was found for sex-related DMCs. This raises the hypothesis that G4s play a regulatory role in escape from X chromosome inactivation.
Время проведения: 18:00-19:30.
28.10.20 [online] Recognition of DNA Secondary Structures as Nucleosome Barriers with Deep Learning Methods.
Совместный семинар с Integrative Biology Group is a part of the Bioengineering Department at the Faculty of Biology of Moscow State Lomonosov University (https://intbio.org/).
Докладчик - Федор Павлов (Факультет компьютерных наук, ВШЭ)
Abstract - Over the past few years, genome research using machine and in-depth learning techniques has become increasingly popular, and researchers are being provided with sophisticated data analysis tools. Recognition of patterns of DNA secondary structures and genomic functional elements are still poorly investigated, despite the fact that research in this area has the potential to contribute greatly to the development of medicine and pharmacology. This study aims to explore machine and deep learning methods that have proven to be successful in natural language processing with respect to the task of DNA sequence recognition. Two deep learning models based on CNN and LSTM architectures were developed. Each model was tested on multiple classification tasks for recognition of DNA sequences containing quadruplexes with potential function of nucleosome barriers. Additionally, model interpretation analysis was performed in the form of extraction of CNN significant filters and their transformation into DNA-motifs.
Время проведения: 16:00-18:00
29.09.20 Разработка лекарственных средств: Как математическое моделирование меняет парадигму разработки новых лекарств в онкологии
Докладчик - Кирилл Песков – кандидат биологических наук, директор ООО «M&S Decisions», руководитель научной группы компьютерной онкологии в Первом МГМУ имени И.М. Сеченова.
Мировой опыт показывает, что использование различных методов компьютерных наук становится неотъемлемой частью процесса разработки оригинальных лекарств. Например, математическое моделирование фармакологических данных широко используются при регистрации лекарственных средств регуляторными органами США и Европейского союза, FDA и EMA, а методы машинного обучения активно востребованы при поиске мишеней и оптимальной структуры химических соединений. В рамках лекции Кирилл Песков расскажет о том, что такое фармакометрика - научная дисциплина о количественном анализе данных клинических исследований и как новые научные методы меняют парадигму разработки лекарств.
Покровский бульвар, 11, корпус R, аудитория R401.
Время проведения: 17:00-18:30
Нашли опечатку?
Выделите её, нажмите Ctrl+Enter и отправьте нам уведомление. Спасибо за участие!
Сервис предназначен только для отправки сообщений об орфографических и пунктуационных ошибках.